


 النبات
النبات
 الحيوان
الحيوان
 الأحياء المجهرية
الأحياء المجهرية
 علم الأمراض
علم الأمراض
 التقانة الإحيائية
التقانة الإحيائية
 التقنية الحيوية المكروبية
التقنية الحيوية المكروبية
 التقنية الحياتية النانوية
التقنية الحياتية النانوية
 علم الأجنة
علم الأجنة
 الأحياء الجزيئي
الأحياء الجزيئي
 علم وظائف الأعضاء
علم وظائف الأعضاء
 الغدد
الغدد
 المضادات الحيوية
المضادات الحيوية|
Read More
Date: 10-5-2016
Date: 10-12-2020
Date: 11-5-2016
|
Frameshifting
Frameshifting is a change in reading frame during gene expression. Transcription errors, or a posttranscriptional process such as RNA Editing, can cause frameshifting. For example, a fraction of the b- amyloid precursor protein and ubiquitin B messenger RNAs in the lesions of Alzheimer's disease are subject to transcriptional frameshifting (1). Most of the frameshifting cases, however, occur during translation (2).
The reading frame is accurately maintained during translation. The average occurrence of frameshift errors is estimated to be 5×10–5 per codon or lower. Aminoacyl tRNA imbalance, due to amino acid starvation or mutations in translation factors or aminoacyl-tRNA synthetases, elevates the error rate. A class of suppressor tRNAs increases frameshifting at particular codons (3). In these cases, the effects are nonspecific. On the other hand, a minority of mRNAs have specific sites for frameshifting that is essential to synthesize the correct gene product. Such natural frameshifting is genetically programmed by specific signals on the mRNAs and, in some cases, is highly efficient (up to 50%. (
Almost all the known programmed frameshifting is either +1 (reading frame shifts to the 5′ direction by one nucleotide) or –1 frameshifting (to the 3′ direction by one nucleotide). An extreme exception is bypassing of 50 nucleotides within the coding region of T4 phage gene 60. The most crucial signal is the sequence of the frameshift site itself. Frameshift sites are often accompanied by a cis-acting sequence element (or stimulator) located either 3′ or 5′ to the frameshift site. Stem-loop or pseudoknot structures of RNA and the Shine–Dalgarno sequence are known to function as 3′ and 5′ stimulators, respectively (Fig. 1). In some cases, a frameshift site itself contains a rare codon or a stop codon as a shift-site stimulator. The basic function of these stimulators is to pause the elongating ribosome to trigger frameshifting. Natural frameshifting stands as a class of reprogrammed genetic decoding (or recoding).
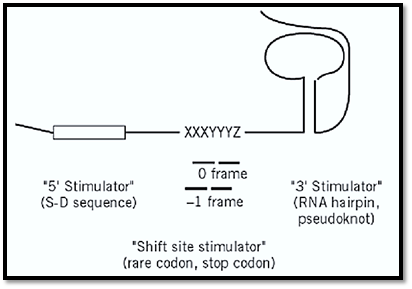
Figure 1. –1 frameshift site motif and stimulators.
Most frameshifting has been found in virus genomes and mobile genetic elements. The former includes several retroviruses such as human immunodeficiency virus 1 (HIV-1) and plant RNA viruses, as well as bacteriophages such as MS2, T7, and l, while the latter includes yeast retrotransposons Ty1 and Ty3, and bacterial insertion sequences (Table 1). In many cases, frameshifting is required for the expression of polymerases (including reverse transcriptase and RNA-dependent RNA polymerase). Some retroviruses, such as human T-cell leukemia virus 1 (HTLV-1) or mouse mammary tumor virus (MMTV), require two frameshifting events to access the reverse transcriptase reading frame. Retroviral frameshifting appears to be a mechanism to keep the synthesis rate of the functional proteins small relative to that of the structural proteins. Although the efficiencies of these frameshifting events are different from one case to another, proper efficiencies are important for optimal viral propagation or transposition in the cases investigated. In viruses and mobile elements, –1 frameshifting is predominant, whereas some viruses and the yeast retrotransposons have +1 frameshift sites.
Table 1. Natural Frameshifting in Wild-Type Genes (2)
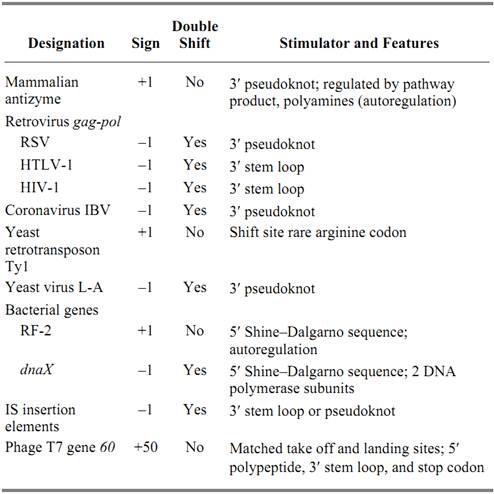
There are only a few instances of frameshifting in bona fide cellular genes. In bacteria, two chromosomal genes, prfB encoding release factor 2 (RF-2; see Release Factor) and Escherichia coli dnaX encoding the g subunit of DNA polymerase III, are expressed by frameshifting. The reading frame of RF-2 switches to the +1 frame, while DnaX shifts to the –1 frame. In higher eukaryotes, there is only one cellular gene known thus far to have frameshifting, the gene for antizyme that is a regulatory protein of the cellular polyamines. Both isoforms of antizyme (AZ1 and AZ2) are expressed by +1 frameshifting that is stimulated by polyamines. RF-2 and antizyme frameshifting is involved in the specific autoregulation (4, 5. (
A majority of –1 frameshift sites are characteristic heptamers with a consensus sequence of X XXW WWY (presented as a preshift reading frame; X and Y are one of any nucleotides and W is either A or U). In addition, most of these –1 shift sites are accompanied by either pseudoknot or stem-loop
structures 3′ to the frameshift site. A simultaneous shift model has been proposed for the shift mechanism, in which the ribosome and the two tRNAs in the P and A sites slip back on the mRNA by one nucleotide, forming new base paring between the tRNAs and the mRNA. Downstream RNA structures stimulate the process by, at least in part, causing ribosomal pausing at the frameshift site (Fig. 1. (
Analyses of +1 frameshifting have revealed that their mechanisms are not as uniform as –1 frameshifting. The codon 3′ adjacent to the frameshift site is either a termination codon or a “hungry” codon [a codon decoded by low-abundant tRNA and thus having low efficiency (6)]. In at least two cases, tRNA slipping and pairing at the new position are not necessary.
Frameshifting can be hinted from open reading frame analysis of complementary DNA sequences or by a disagreement between the sequences of cDNA and its protein product. It is, however, sometimes difficult to prove before knowing the amino acid sequence of the protein. Frameshifting can be induced by expression of exotopic genes or artificial sequences. It is noteworthy that translational frameshifting, even with a low efficiency, can partially rescue frameshift mutations and modifies the phenotype in both experimental and naturally occurring systems, demonstrating the biological importance of frameshifting.
References
1. F. W. van Leeuwen et al. (1998) Science 279, 242–247.
2. J. F. Atkins and R. F. Gesteland (1995) In tRNA: Structure, Biosynthesis, and Function (D. Söll and U. L. RajBhandary, eds.), American Society of Microbiology Press, Washington, D.C., pp. 471-490.
3. E. J. Murgola (1995) In tRNA: Structure, Biosynthesis, and Function (D. Söll and U. L. RajBhandary, eds.), American Society of Microbiology Press, Washington, D.C., pp. 491–509.
4. W. J. Craigen, R. G. Cook, W. P. Tate, and C. T. Caskey (1985) Proc. Natl. Acad. Sci. USA 82, 3616-3620.
5. S. Matsufuji et al. (1995) Cell 80, 51–60.
6. K. Kawakami et al. (1993) Genetics 135, 309–320.

|
|
|
|
دراسة: إجراء واحد لتقليل المخاطر الجينية للوفاة المبكرة
|
|
|
|
|
|
|
"الملح والماء" يمهدان الطريق لأجهزة كمبيوتر تحاكي الدماغ البشري
|
|
|
|
|
|
بالصور: عند زيارته لمعهد نور الإمام الحسين (عليه السلام) للمكفوفين وضعاف البصر في كربلاء.. ممثل المرجعية العليا يقف على الخدمات المقدمة للطلبة والطالبات
|
|
|
|
ممثل المرجعية العليا يؤكد استعداد العتبة الحسينية لتبني إكمال الدراسة الجامعية لشريحة المكفوفين في العراق
|
|
|
|
ممثل المرجعية العليا يؤكد على ضرورة مواكبة التطورات العالمية واستقطاب الكفاءات العراقية لتقديم أفضل الخدمات للمواطنين
|
|
|
|
العتبة الحسينية تستملك قطعة أرض في العاصمة بغداد لإنشاء مستشفى لعلاج الأورام السرطانية ومركز تخصصي للتوحد
|