النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الميكروبية

مواضيع عامة في المضادات الميكروبية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
Some Group I Introns Encode Endonucleases That Sponsor Mobility
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المصدر:
LEWIN’S GENES XII
الجزء والصفحة:
21-5-2021
2421
Some Group I Introns Encode Endonucleases That Sponsor Mobility
KEY CONCEPTS
- Mobile introns are able to insert themselves into new sites.
- Mobile group I introns encode an endonuclease that makes a double-strand break at a target site.
- The intron transposes into the site of the double-strand break by a DNA-mediated replicative mechanism.
Certain introns of both the group I and group II classes contain open reading frames that are translated into proteins. Expression of the proteins allows the intron (either in its original DNA form or as a DNA copy of the RNA) to be mobile: It is able to insert itself into a new genomic site. Introns of groups I and II are widespread, being found in both prokaryotes and eukaryotes. Group I introns migrate by DNA-mediated mechanisms, whereas group II introns migrate by RNA-mediated mechanisms.
Intron mobility was first detected by crosses in which the alleles for the relevant gene differ with regard to the presence of the intron. Polymorphisms for the presence or absence of introns are common in fungal mitochondria. This is consistent with the view that these introns originated by insertion into the gene. Some light on the process that could be involved is cast by an analysis of recombination in crosses involving the large rRNA gene of the yeast mitochondrion.
The large rRNA gene of the yeast mitochondrion has a group I intron that contains a coding sequence. The intron is present in some strains of yeast (called ω+ ) but absent in others (ω- ).
Progeny of genetic crosses between ω+ and ω- do not result in the expected genotypic ratio; the progeny are usually ω+ . If we think of the ω+ strain as a donor and the ω- strain as a recipient, we form the view that in ω+ × ω- crosses a new copy of the intron is generated in the ω- genome. As a result, all of the progeny are ω+ . Mutations can occur in either parent to abolish the non-Mendelian genotypic assortment. Certain mutants show normal segregation, with equal numbers of ω+ and ω- progeny. When mapped, mutations in the ω- strain occur close to the site where the intron would be inserted. Mutations in the ω+ strain lie in the reading frame of the intron and prevent production of the protein. This suggests the model shown in FIGURE 1, in which the protein encoded by the intron in an ω+ strain recognizes the site where the intron should be inserted into an ω strain and causes it to be preferentially inherited.
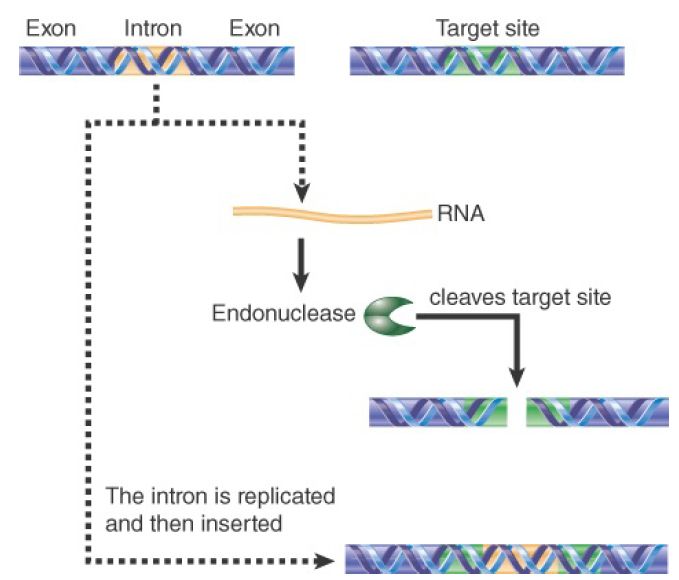
FIGURE 1.An intron encodes an endonuclease that makes a double-strand break in DNA. The sequence of the intron is duplicated and then inserted at the break.
Some group I introns encode endonucleases that make them mobile. At least six families of homing endonuclease genes (HEGs) have been identified. Two common families of HEGs are the LAGLIDADG and His-Cys box endonucleases. However, these HEG-containing group I introns constitute a small portion of the overall number of group I introns.
The ω intron contains an HEG, the product of which is an endonuclease known as I-SceI. I-SceI recognizes the ω- gene as a target for a double-strand break. I-SceI recognizes an 18-bp
target sequence that contains the site where the intron is inserted.
The target sequence is cleaved on each strand of DNA two bases to the 3′ side of the insertion site. Thus, the cleavage sites are 4 bp apart and generate overhanging single strands. This type of cleavage is related to the cleavage characteristic of transposons when they migrate to new sites . The double-strand break probably initiates a gene conversion process in which the sequence of the ω gene is copied to replace the sequence of the ω gene. The reaction involves transposition by a duplicative mechanism and occurs solely at the level of DNA. Insertion of the intron interrupts the sequence recognized by the endonuclease, thus ensuring stability. (Homing endonucleases have also been adapted for use in genome editing technologies.)
Similar introns often carry quite different endonucleases. The details of insertion differ; for example, the endonuclease encoded by the phage T4 td intron cleaves a target site that is 24 bp upstream of the site at which the intron is itself inserted. The dissociation between the intron sequence and the endonuclease sequence is emphasized by the fact that the same endonuclease
sequences are found in inteins (sequences that encode self-splicing proteins).
The variation in the endonucleases means that there is no homology between the sequences of their target sites. The target sites are among the longest, and therefore the most specific, known for any endonucleases (with a range of 14 to 40 bp). The specificity ensures that the intron perpetuates itself only by insertion into a single target site and not elsewhere in the genome. This is called intron homing.
Introns carrying sequences that encode endonucleases are found in a variety of bacteria and unicellular/oligocellular eukaryotes. These results strengthen the view that introns carrying coding sequences originated as independent elements.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة

الآخبار الصحية















 قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة
قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة "المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة
"المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة (نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)
(نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)